Voy a empezar este post con un par de citas.
El análisis de datos es básicamente encontrar la matriz correcta a diagonalizar.
Quien renuncia a la estructura, deja dinero encima de la mesa.
La primera no recuerdo dónde la leí, pero es de la escuela francesa de estadística, la segunda es del blog hermano datanalytics.
Y bueno, ambas tienen parte de razón. En esta entrada voy a comentar brevemente una forma de imputación de datos que quizá a alguien le sea útil.
La idea básica es:
- Imputar los valores perdidos por la media de cada variable.
- Calcular la estructura factorial con el dataset anterior.
- Imputar los valores perdidos por los valores predichos por la estructura factorial. Usando la matriz X reconstruida.
- Repetir hasta convergencia.
Este procedimiento iterativo suele usar Expectation Maximization y se conoce como EM-PCA. (también hay versión usando regularización)
Más información en este artículo de François Husson
Ejemplo
Y ahora vamos a ver un ejemplito, para eso vamos a usar la librería missMDA de François Husson y Julie Josse, que incorpora estos métodos.
library(missMDA)Usamos el conjunto de datos orange en cuya ayuda leeemos
Description Sensory description of 12 orange juices by 8 attributes. Some values are missing.
Usage data(orange) Format A data frame with 12 rows and 8 columns. Rows represent the different orange juices, columns represent the attributes.
data(orange)Por ejemplo, tenemos un valor perdido para la variable Attack.intensity en la primera fila
DT::datatable(orange,
options = list(scrollX = TRUE))Bien, pues ahora podemos usar el EM-PCA para imputar los valores perdidos teniendo en cuenta la estructura factorial. Usamos el ejemplo que viene en la función.
Estimamos el número de componentes a extraer y usamos la función imputePCA que
estim_ncpPCA(orange)## Warning in impute(X, ncp = ncp, scale = scale, method = method, ind.sup =
## ind.sup, : Stopped after 1000 iterations## $ncp
## [1] 2
##
## $criterion
## 0 1 2 3 4 5
## 1.0388714 0.9279948 0.5976727 0.7855501 2.0250470 2.6741735res_impute <- imputePCA(orange, ncp=2)La función devuelve el conjunto de datos ya imputado
res_impute$completeObs## Color.intensity Odor.intensity Attack.intensity Sweet Acid Bitter
## 1 4.791667 5.291667 4.077034 5.527352 4.177564 2.833333
## 2 4.583333 6.041667 4.416667 5.458333 4.125000 3.541667
## 3 4.708333 5.333333 4.158054 5.442936 4.291667 3.166667
## 4 6.583333 6.000000 7.416667 4.166667 6.750000 4.702509
## 5 6.271605 6.166667 5.333333 4.083333 5.455805 4.375000
## 6 6.333333 5.000000 5.375000 5.000000 5.500000 3.625000
## 7 4.291667 4.916667 5.291667 5.541667 5.250000 3.214232
## 8 4.460613 4.541667 4.833333 5.479128 4.958333 2.916667
## 9 4.416667 5.136550 5.166667 4.625000 5.041667 3.666667
## 10 4.541667 4.291667 4.176991 5.791667 4.375000 2.735255
## 11 4.083333 5.125000 3.916667 5.703297 3.900164 2.815857
## 12 6.500000 5.875000 6.125000 4.875000 5.291667 4.166667
## Pulp Typicity
## 1 5.711715 5.208333
## 2 4.625000 4.458333
## 3 6.250000 5.166667
## 4 1.416667 3.416667
## 5 3.416667 4.416667
## 6 4.208333 4.875000
## 7 1.291667 4.333333
## 8 1.541667 3.958333
## 9 1.541667 3.958333
## 10 4.026062 5.000000
## 11 7.333333 5.250000
## 12 1.500000 3.500000Extensiones
Se puede hacer imputación múltiple.
# creamos 100 datasets imputados,
# por defecto usa método bootstrap pero también puede usar una versión bayesiana
res_mi_impute <- MIPCA(orange, ncp = 2, nboot = 100)En el Slot res.MI tenemos los datasets imputados
# vemos por ejemplo la imputación 3
res_mi_impute$res.MI[[3]]## Color.intensity Odor.intensity Attack.intensity Sweet Acid Bitter
## 1 4.791667 5.291667 3.954364 5.879781 4.656048 2.833333
## 2 4.583333 6.041667 4.416667 5.458333 4.125000 3.541667
## 3 4.708333 5.333333 4.333241 6.212547 4.291667 3.166667
## 4 6.583333 6.000000 7.416667 4.166667 6.750000 4.963685
## 5 5.513219 6.166667 5.333333 4.083333 5.867583 4.375000
## 6 6.333333 5.000000 5.375000 5.000000 5.500000 3.625000
## 7 4.291667 4.916667 5.291667 5.541667 5.250000 2.782590
## 8 4.741117 4.541667 4.833333 4.519020 4.958333 2.916667
## 9 4.416667 5.030443 5.166667 4.625000 5.041667 3.666667
## 10 4.541667 4.291667 4.282674 5.791667 4.375000 2.442581
## 11 4.083333 5.125000 3.916667 6.010708 3.530739 2.929682
## 12 6.500000 5.875000 6.125000 4.875000 5.291667 4.166667
## Pulp Typicity
## 1 8.327411 5.208333
## 2 4.625000 4.458333
## 3 6.250000 5.166667
## 4 1.416667 3.416667
## 5 3.416667 4.416667
## 6 4.208333 4.875000
## 7 1.291667 4.333333
## 8 1.541667 3.958333
## 9 1.541667 3.958333
## 10 5.029165 5.000000
## 11 7.333333 5.250000
## 12 1.500000 3.500000Cuando el conjunto de datos es pequeño, como en este caso (12 filas), podemos ver gráficamente la incertidumbre asociada a la imputación. En la ayuda de la función plot.MIPCA se puede consultar lo que significa cada gráfico
plot(res_mi_impute)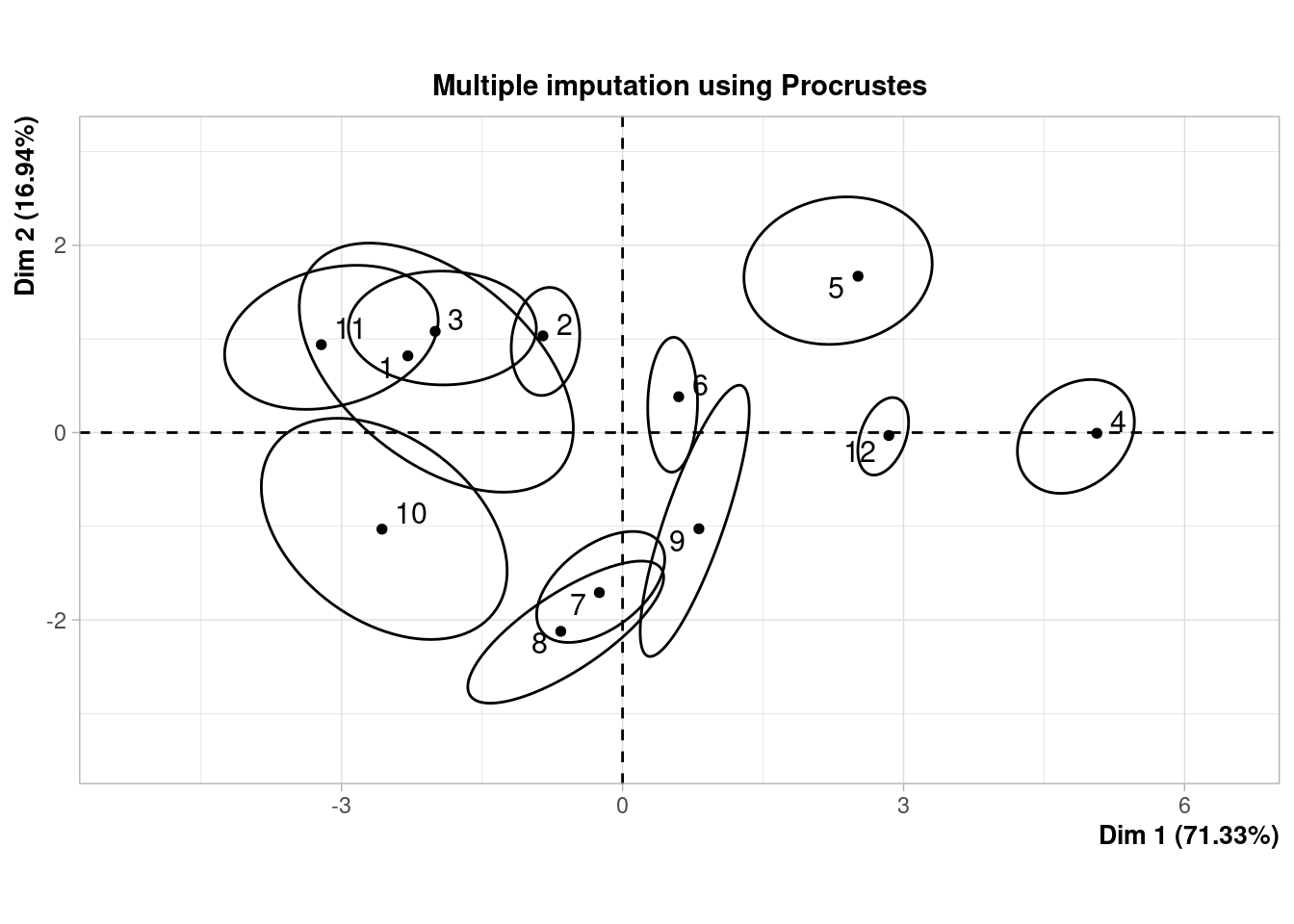
## $PlotIndProc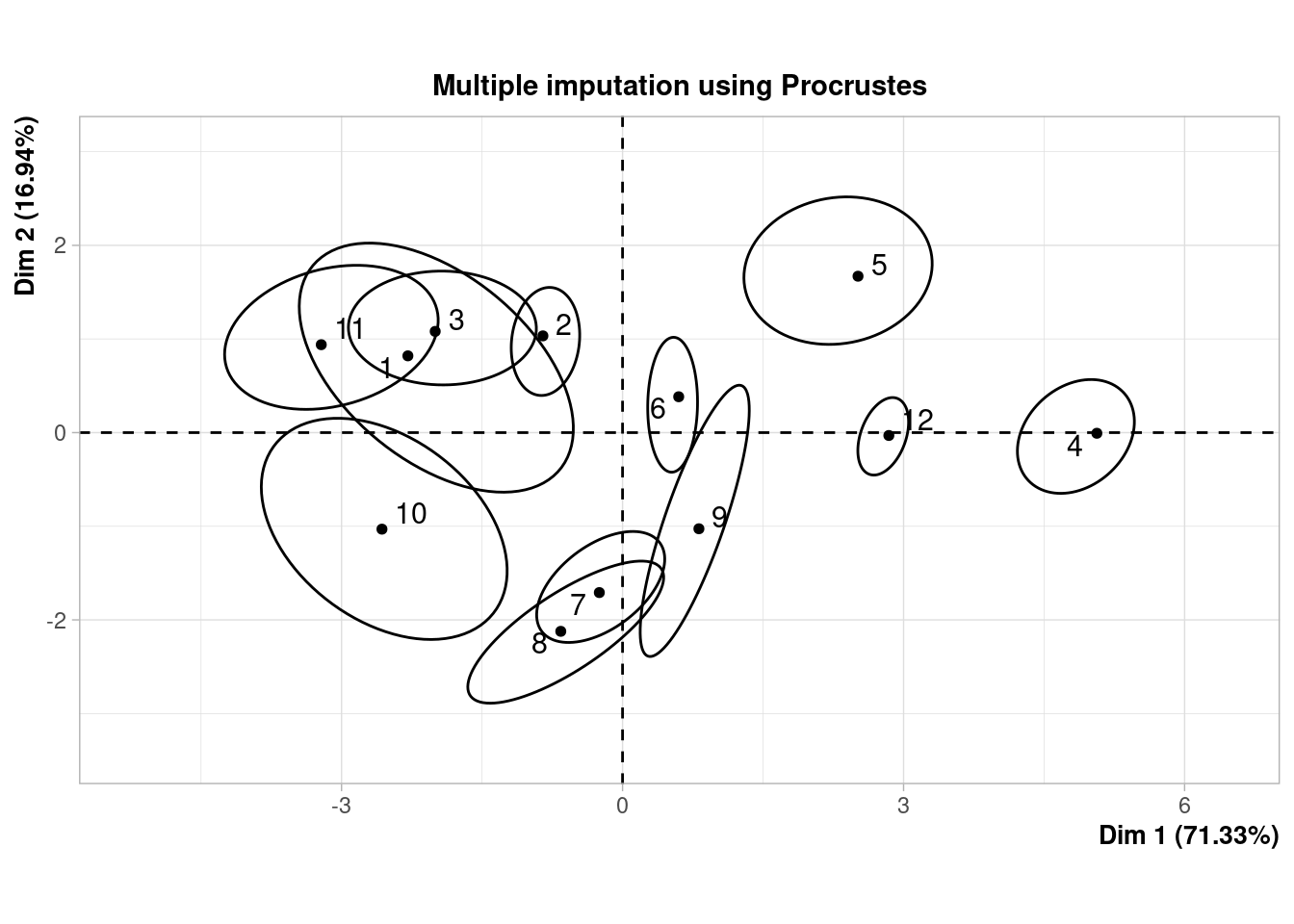
##
## $PlotDim
##
## $PlotIndSupp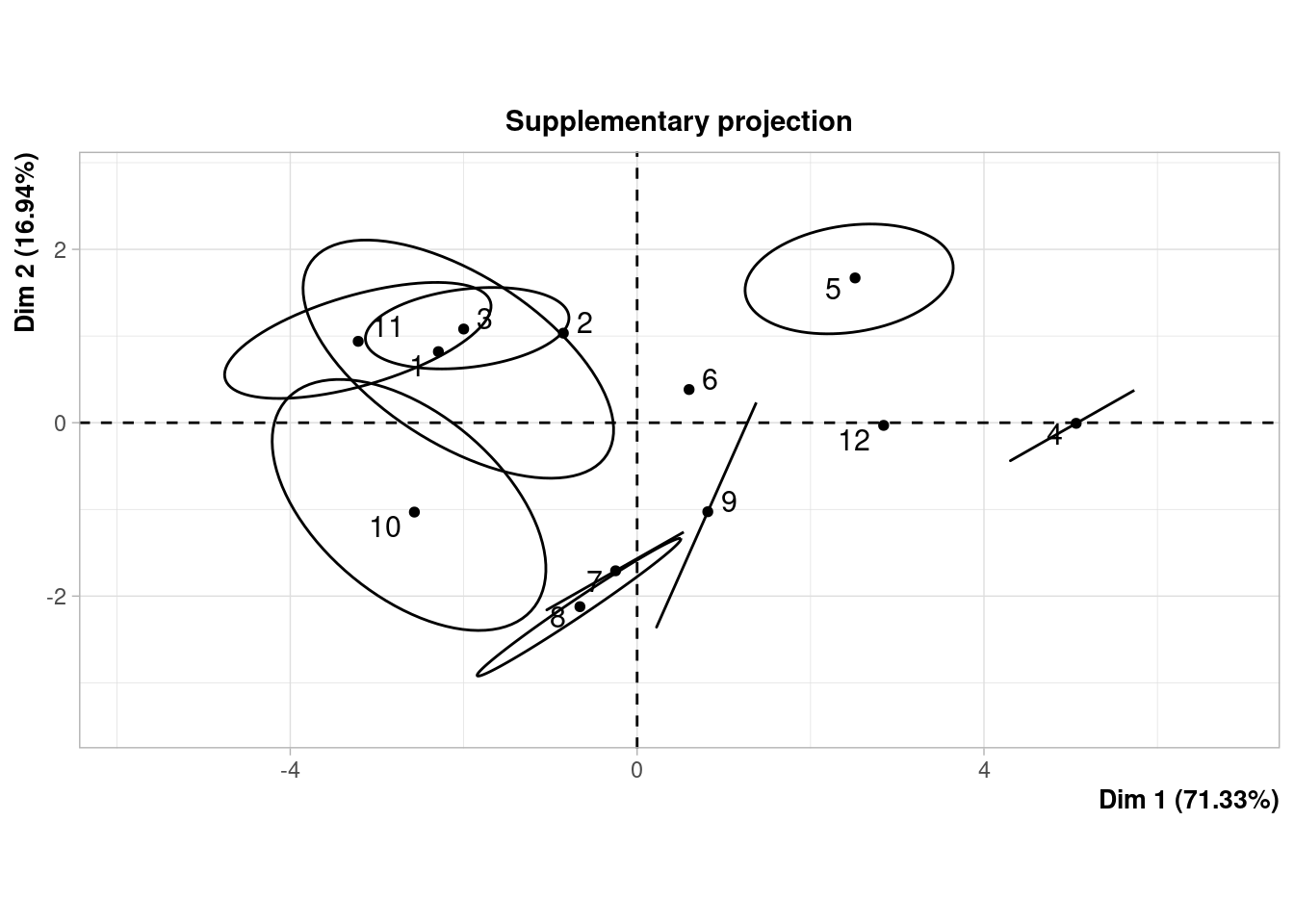
##
## $PlotVar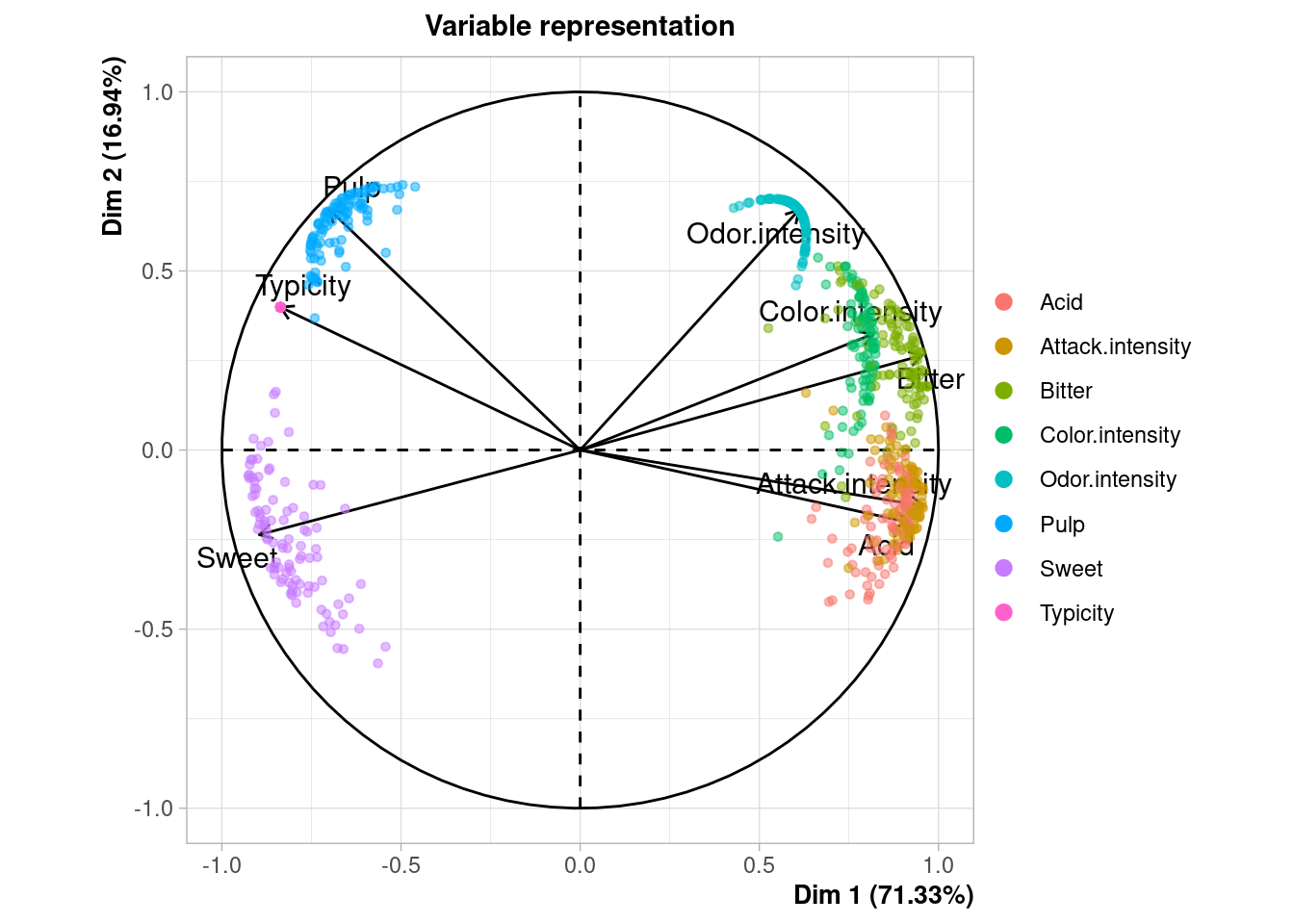
Mas extensiones
La librería que subyace bajo missMDA es la conocida FactoMineR que permite hacer métodos factoriales (otros nombres para lo mismo serían métodos de componentes principales, reducción de dimensiones, projection pursuit) teniendo en cuenta que las variables sean categóricas (Análisis de correspondencias simple y múltiple) o que haya una mezcla de continuas y categóricas (Análisis factorial para datos mixtos).
Una de las extensiones más útiles a mi modo de ver es la que permite imputar teniendo en cuenta la estructura factorial y también que las observaciones estén asociadas, por ejemplo que tenga una clasificación previa, vía segmentación o un cluster previo.
Por ejemplo podría tener datos de clientes de diferentes provincias de España, unirlo todo en un conjunto de datos dónde tenga la variable que indica de qué provincia es cada cliente y poder obtener una estructura factorial global general y una estructura factorial específica para cada provincia y poder utilizar ambas estructuras para imputar los valores perdidos. Sería una parte general y una parte específica. ¿No os recuerda a los modelos mixtos bayesianos o a la estrategia de de modelo global y particular de Carlos ?
Bueno, pues todo eso se puede hacer con la librería missMDA. Como ejemplo podemos ver el dataset ozone
Description This dataset contains 112 daily measurements of meteorological variables (wind speed, temperature, rainfall, etc.) and ozone concentration recorded in Rennes (France) during the summer 2001. There are 11 continuous variables and 2 categorical variables with 2 or 4 levels. Some values are missing.
data(ozone)
head(ozone,6)## maxO3 T9 T12 T15 Ne9 Ne12 Ne15 Vx9 Vx12 Vx15 maxO3v
## 20010601 87 15.6 18.5 18.4 4 4 8 0.6946 -1.7101 -0.6946 84
## 20010602 NA 17.0 18.4 17.7 5 5 7 -4.3301 -4.0000 -3.0000 87
## 20010603 92 15.3 17.6 19.5 2 5 4 2.9544 1.8794 0.5209 82
## 20010604 114 16.2 19.7 22.5 1 NA 0 0.9848 NA NA 92
## 20010605 94 17.4 20.5 20.4 8 8 7 -0.5000 -2.9544 -4.3301 114
## 20010606 80 17.7 19.8 18.3 6 6 7 -5.6382 -5.0000 -6.0000 NA
## vent pluie
## 20010601 Nord Sec
## 20010602 Nord Sec
## 20010603 Est <NA>
## 20010604 <NA> Sec
## 20010605 Ouest Sec
## 20010606 Ouest PluieY vamos a usar como variable de grupo vent y vemos que tenemos por ejemplo paara la segunda fila valores perdidos en la variable numérica maxO3 o para la tercer fila tenemos un valor perdido para la variable categórica pluie
ozone[c('20010602','20010603'), ]## maxO3 T9 T12 T15 Ne9 Ne12 Ne15 Vx9 Vx12 Vx15 maxO3v vent
## 20010602 NA 17.0 18.4 17.7 5 5 7 -4.3301 -4.0000 -3.0000 87 Nord
## 20010603 92 15.3 17.6 19.5 2 5 4 2.9544 1.8794 0.5209 82 Est
## pluie
## 20010602 Sec
## 20010603 <NA>Para aplicar el método de imputación tenemos que decidir qué número de componentes queremos para la estructura general y cuántos para la particular.
# en ifac tenemos que poner el índice de la variable de grupo (de tipo factor), vent es la 12
ncp_estim <- estim_ncpMultilevel(ozone, ifac = 12)Y nos devuelve el número de componentes estimados entre los grupos ncpB y el número de componentes estimados dentro de los grupos.
ncp_estim$ncpB## [1] 1ncp_estim$ncpW## [1] 3Y con esto se lo pasamos a la función imputeMultilevel y hacemos la imputación
ozone_multilevel_imp <- imputeMultilevel(ozone, ifac = 12, ncpB = 1, ncpW= 3)## Warning in imputeMultilevel(ozone, ifac = 12, ncpB = 1, ncpW = 3): Rows with
## missing valued on the group variables are droppedY podemos ver la imputación que ha realizado
head(ozone_multilevel_imp$completeObs)## maxO3 T9 T12 T15 Ne9 Ne12 Ne15 Vx9 Vx12 Vx15
## 20010601 87.00000 15.6 18.5 18.4 4 4 8 0.6946 -1.7101 -0.6946
## 20010602 79.51042 17.0 18.4 17.7 5 5 7 -4.3301 -4.0000 -3.0000
## 20010603 92.00000 15.3 17.6 19.5 2 5 4 2.9544 1.8794 0.5209
## 20010605 94.00000 17.4 20.5 20.4 8 8 7 -0.5000 -2.9544 -4.3301
## 20010606 80.00000 17.7 19.8 18.3 6 6 7 -5.6382 -5.0000 -6.0000
## 20010607 68.07716 16.8 15.6 14.9 7 8 8 -4.3301 -1.8794 -3.7588
## maxO3v vent pluie
## 20010601 84.00000 Nord Sec
## 20010602 87.00000 Nord Sec
## 20010603 82.00000 Est Sec
## 20010605 114.00000 Ouest Sec
## 20010606 75.52026 Ouest Pluie
## 20010607 80.00000 Ouest SecY vemos que ha realizado tanto imputación de la variable numérica maxO3 para el dato de 20010602, como imputación de la variable categórica pluie para el datos de 20010603
ozone_multilevel_imp$completeObs[c('20010602','20010603'), c("maxO3", "pluie") ]## maxO3 pluie
## 20010602 79.51042 Sec
## 20010603 92.00000 SecLo dicho, me parece una técnica interesante cuándo se tiene por ejemplo una variable que te clasifica los clientes (ya sea una clasificación previa o dada por negocio) y tenemos datos faltantes tanto para variables numéricas como categóricas. Me parece una mejor solución que imputar por la media, mediana o simplemente asignar un valor pseudo aleatorio tipo -9999. Además de que el enfoque geométrico-algebraico de las técnicas de componentes principales siempre me ha gustado.
Y como decía al principio Quien renuncia a la estructura, deja dinero encima de la mesa.
Feliz semana.