Hace un par de días un amigo mío me preguntaba por temas de que quería paralelizar un proceso con R, y no acababa de ver claro cómo. A falta de que mande un ejemplo creí entender que tiene un dataframe dónde tiene un proceso que encuentra para cada fila un conjunto de n filas parecidas y sobre ese conjunto de n filas hace cosas, como estimar algo y tal. Y lo que quiere al final es tener tanto lo estimado como un dataframe con las n filas similares a cada fila original
Bueno, hasta que me mande el ejemplo y me entere bien, me acordé que en R y más concretamente en el mundo tidyverse tenemos los nested data, que nos permite tener columnas cuyos elementos pueden ser cualquier cosa, desde un json, un dataframe, un modelo , etcc..
Veamos un ejemplo
purrr
library(tidyverse)## ── Attaching packages ─────────────────────────────────────── tidyverse 1.3.0 ──## ✓ ggplot2 3.3.3 ✓ purrr 0.3.4
## ✓ tibble 3.1.0 ✓ dplyr 1.0.4
## ✓ tidyr 1.1.2 ✓ stringr 1.4.0
## ✓ readr 1.4.0 ✓ forcats 0.5.1## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()df_nest <- tibble(
grupo = c(letters[1:5]),
dfs = list(
dfa = data.frame(x = rnorm(2000), y = rexp(2000)),
dfb = data.frame(x = rnorm(1000), y = rexp(1000)),
dfc = data.frame(x = rnorm(30), y = rexp(30)),
dfd = data.frame(x = rnorm(200), y = rexp(200)),
dfe = data.frame(x = rnorm(1e5), y = rexp(1e5))
)
)
df_nest## # A tibble: 5 x 2
## grupo dfs
## <chr> <named list>
## 1 a <df[,2] [2,000 × 2]>
## 2 b <df[,2] [1,000 × 2]>
## 3 c <df[,2] [30 × 2]>
## 4 d <df[,2] [200 × 2]>
## 5 e <df[,2] [100,000 × 2]>Y vemos que el primer elemento de la columna dfs es un dataframe de 2000 filas y 2 colmnas y que el quinto tiene 100 mil filas de filas y dos columnas. Esta forma de tener la información puede ser útil, o al menos a mi me lo parece.
¿Podríamos ahora hacer, por ejemplo un modelo sobre cada elemento de dfs? Si, y de manera muy sencilla utilizando funciones de purrr ya incluida con tidyverse
df_con_modelo <- df_nest %>%
mutate(modelo = map(dfs, function(df)
lm(y ~ x, df))) %>%
mutate(resumen_modelo = map(modelo, broom::tidy)) # añodo tambin tabla resumen
df_con_modelo## # A tibble: 5 x 4
## grupo dfs modelo resumen_modelo
## <chr> <named list> <named list> <named list>
## 1 a <df[,2] [2,000 × 2]> <lm> <tibble [2 × 5]>
## 2 b <df[,2] [1,000 × 2]> <lm> <tibble [2 × 5]>
## 3 c <df[,2] [30 × 2]> <lm> <tibble [2 × 5]>
## 4 d <df[,2] [200 × 2]> <lm> <tibble [2 × 5]>
## 5 e <df[,2] [100,000 × 2]> <lm> <tibble [2 × 5]>Y podemos ver los modelos o los resúmenes
df_con_modelo$modelo[[2]]##
## Call:
## lm(formula = y ~ x, data = df)
##
## Coefficients:
## (Intercept) x
## 0.97521 -0.08025# o equivalentemente
df_con_modelo %>%
pull(modelo) %>%
.[[5]]##
## Call:
## lm(formula = y ~ x, data = df)
##
## Coefficients:
## (Intercept) x
## 1.000044 0.001346df_con_modelo$resumen_modelo[[2]]## # A tibble: 2 x 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 0.975 0.0301 32.4 8.88e-158
## 2 x -0.0802 0.0303 -2.65 8.20e- 3Hagamos un ejemplo más elaborado, usando datos de la librería gapminder más info en la ayuda de la misma
library(gapminder)
DT::datatable(gapminder)Podemos construirnos un dataset con datos agregados por país o por continente usando la función nest
df <- gapminder %>%
group_by(continent) %>%
nest()
df## # A tibble: 5 x 2
## # Groups: continent [5]
## continent data
## <fct> <list>
## 1 Asia <tibble [396 × 5]>
## 2 Europe <tibble [360 × 5]>
## 3 Africa <tibble [624 × 5]>
## 4 Americas <tibble [300 × 5]>
## 5 Oceania <tibble [24 × 5]>Y ahora en data tenemos los datos de cada continente
Ahora el objetivo es hacer un modelo para cada continente un modelo que relacione la esperanza de vida y el año.
Para eso nos creamos un par de funciones, que usaremos en el map. Una para hacer el modelo y otra un gráfico.
make_modelo_simple <- function(df){
modelo_simple <- function(df) {
lm(lifeExp ~ year , df)
}
# el uso de possibly mola.
modelo_safe <- possibly(modelo_simple, otherwise = NA)
return(modelo_safe(df))
}
plot_lifeexp <- function(df){
df %>%
ggplot(aes(x= year, y=lifeExp, color = country, group= country)) +
geom_point() +
geom_line() +
labs(title = "LifeExp evolution")
}Y ahora hacemos un gráfico para cada continente, un modelo por continente y obtenemos las predicciones.
df2 <- df %>%
mutate(
# modelo
modelo_x_continente = map(data, make_modelo_simple),
# grafico
plots = map(data, plot_lifeexp),
# uso de map2 para funcion con dos argumentos
predicciones = map2(modelo_x_continente,
data,
function(modelo, datos)
predict(modelo, newdata = datos)
)
)
df2## # A tibble: 5 x 5
## # Groups: continent [5]
## continent data modelo_x_continente plots predicciones
## <fct> <list> <list> <list> <list>
## 1 Asia <tibble [396 × 5]> <lm> <gg> <dbl [396]>
## 2 Europe <tibble [360 × 5]> <lm> <gg> <dbl [360]>
## 3 Africa <tibble [624 × 5]> <lm> <gg> <dbl [624]>
## 4 Americas <tibble [300 × 5]> <lm> <gg> <dbl [300]>
## 5 Oceania <tibble [24 × 5]> <lm> <gg> <dbl [24]>Y por ejemplo, el plot para Oceanía
df2$plots[[5]]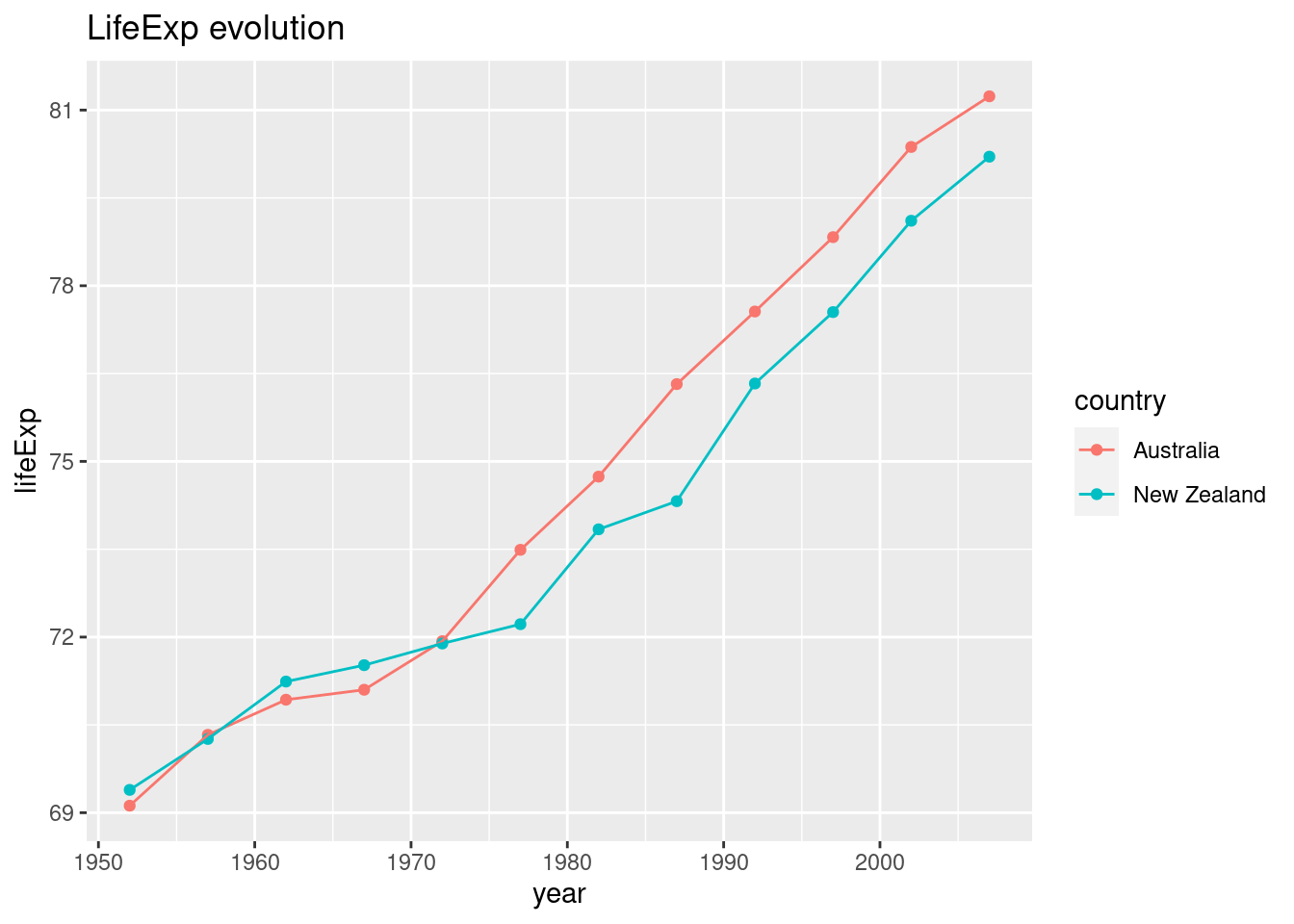
Y su modelo y predicciones
df2$modelo_x_continente[[5]]##
## Call:
## lm(formula = lifeExp ~ year, data = df)
##
## Coefficients:
## (Intercept) year
## -341.9080 0.2103df2$predicciones[[5]]## 1 2 3 4 5 6 7 8
## 68.54372 69.59508 70.64644 71.69780 72.74917 73.80053 74.85189 75.90325
## 9 10 11 12 13 14 15 16
## 76.95461 78.00597 79.05734 80.10870 68.54372 69.59508 70.64644 71.69780
## 17 18 19 20 21 22 23 24
## 72.74917 73.80053 74.85189 75.90325 76.95461 78.00597 79.05734 80.10870furrr
¿Y podemos hacer todo esto en paralelo? Pues si, y muy fácil con la librería furrr que usa la fantástica future. Con future podemos usar múltiples procesadores, múltiples sesiones, o incluso montar un cluster sobre varias máquinas usando MPI (eso si era computación distribuida y no spark , que me lo ha contado mi amigo Rubén, que fue sysadmin en uno de esos sistemas).
Para usar furrr aparte de instalarlo lo que tenemos que especificar es el tipo de plan, yo recomiendo usar plan(multisession) que no da problemas con Rstudio como si da plan(multiprocess)
library(furrr
)## Loading required package: futureplan(multisession, workers = 5) # utilizo 5 workers porque tengo 5 contintentesY para hacer lo mismo que con purrr solo necesitamos añadir el prefijo future a los map del ejemplo anterior y ya se hace en paralelo.
df3 <- df %>%
mutate(
modelo_x_continente = future_map(data,make_modelo_simple),
plots = future_map(data, plot_lifeexp),
predicciones = future_map2( modelo_x_continente,data,
function(modelo,datos)
predict(modelo, newdata = datos))
)
df3## # A tibble: 5 x 5
## # Groups: continent [5]
## continent data modelo_x_continente plots predicciones
## <fct> <list> <list> <list> <list>
## 1 Asia <tibble [396 × 5]> <lm> <gg> <dbl [396]>
## 2 Europe <tibble [360 × 5]> <lm> <gg> <dbl [360]>
## 3 Africa <tibble [624 × 5]> <lm> <gg> <dbl [624]>
## 4 Americas <tibble [300 × 5]> <lm> <gg> <dbl [300]>
## 5 Oceania <tibble [24 × 5]> <lm> <gg> <dbl [24]>df3$plots[[2]]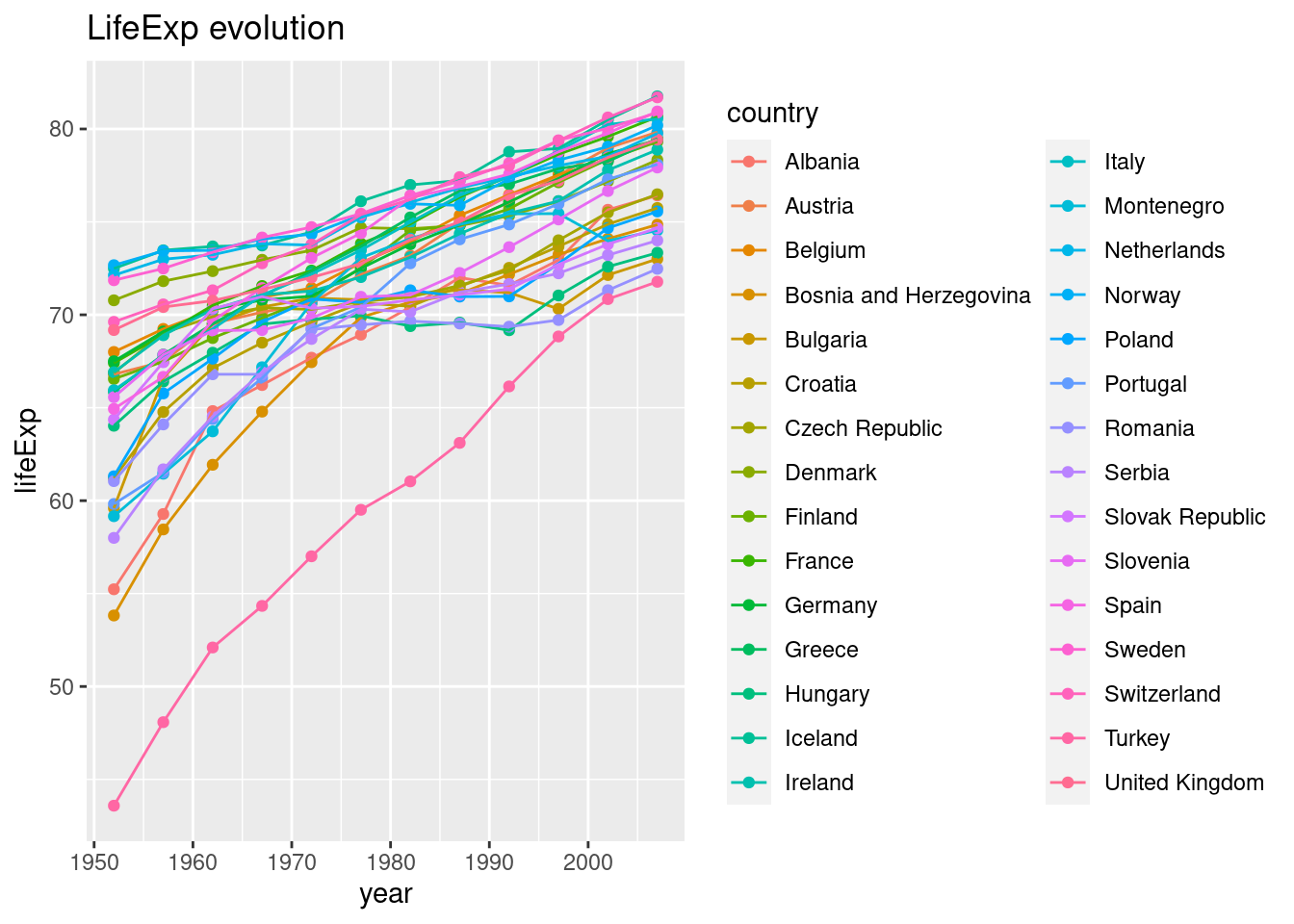
Bueno, espero que le sirva a mi colega.