En estos tiempos tan asépticos ya no estamos acostumbrados (en algunos países), a tratar con agentes patógenos altamente contagiosos como el que llena los titulares de periódicos y televisiones estos días.
Sin más, vamos a comparar los datos de España e Italia, plagiando con total descaro a mi amigo Carlos Gil que puso este post de ayer y en este de hoy.
Yo me acabo de enterar de que estoy en cuarentena preventiva por un posible contagio de la mujer de un compañero, así que en casita a teletrabajar unos días.
El código
# Librerías ----
library(tidyverse)## ── Attaching packages ──────────────────────────────────────── tidyverse 1.3.0.9000 ──## ✓ ggplot2 3.3.0 ✓ purrr 0.3.3
## ✓ tibble 2.1.3 ✓ dplyr 0.8.5
## ✓ tidyr 1.0.2 ✓ stringr 1.4.0
## ✓ readr 1.3.1 ✓ forcats 0.5.0## ── Conflicts ──────────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()library(leaflet)
library(htmlwidgets)
library(htmltools)
# nos bajamos los datos, actualizados a día de ayer ----
url <- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
cvirus <- read.table(url, sep = ",", header = T)
cvirus$Province.State <- NULL
# Pasamos a formato largo ----
cvirus_longer <- cvirus %>%
pivot_longer(
cols = starts_with("X"),
values_to = "casos"
)
colnames(cvirus_longer) <- c("país", "Lat", "Long", "fecha", "casos")
cvirus_longer$fecha <- as.Date(as.character(cvirus_longer$fecha), format = "X%m.%d.%y")
# Nos quedamos con los paises que nos interesan
cvirus_it_sp <- cvirus_longer %>%
filter(país %in% c("Italy", "Spain"))
head(cvirus_it_sp)## # A tibble: 6 x 5
## país Lat Long fecha casos
## <fct> <dbl> <dbl> <date> <int>
## 1 Italy 43 12 2020-01-22 0
## 2 Italy 43 12 2020-01-23 0
## 3 Italy 43 12 2020-01-24 0
## 4 Italy 43 12 2020-01-25 0
## 5 Italy 43 12 2020-01-26 0
## 6 Italy 43 12 2020-01-27 0A pintar.
cvirus_it_sp %>%
filter(fecha > as.Date("2020-02-23")) %>%
ggplot(aes(x = fecha, y = casos, col = país)) +
geom_point() +
geom_line()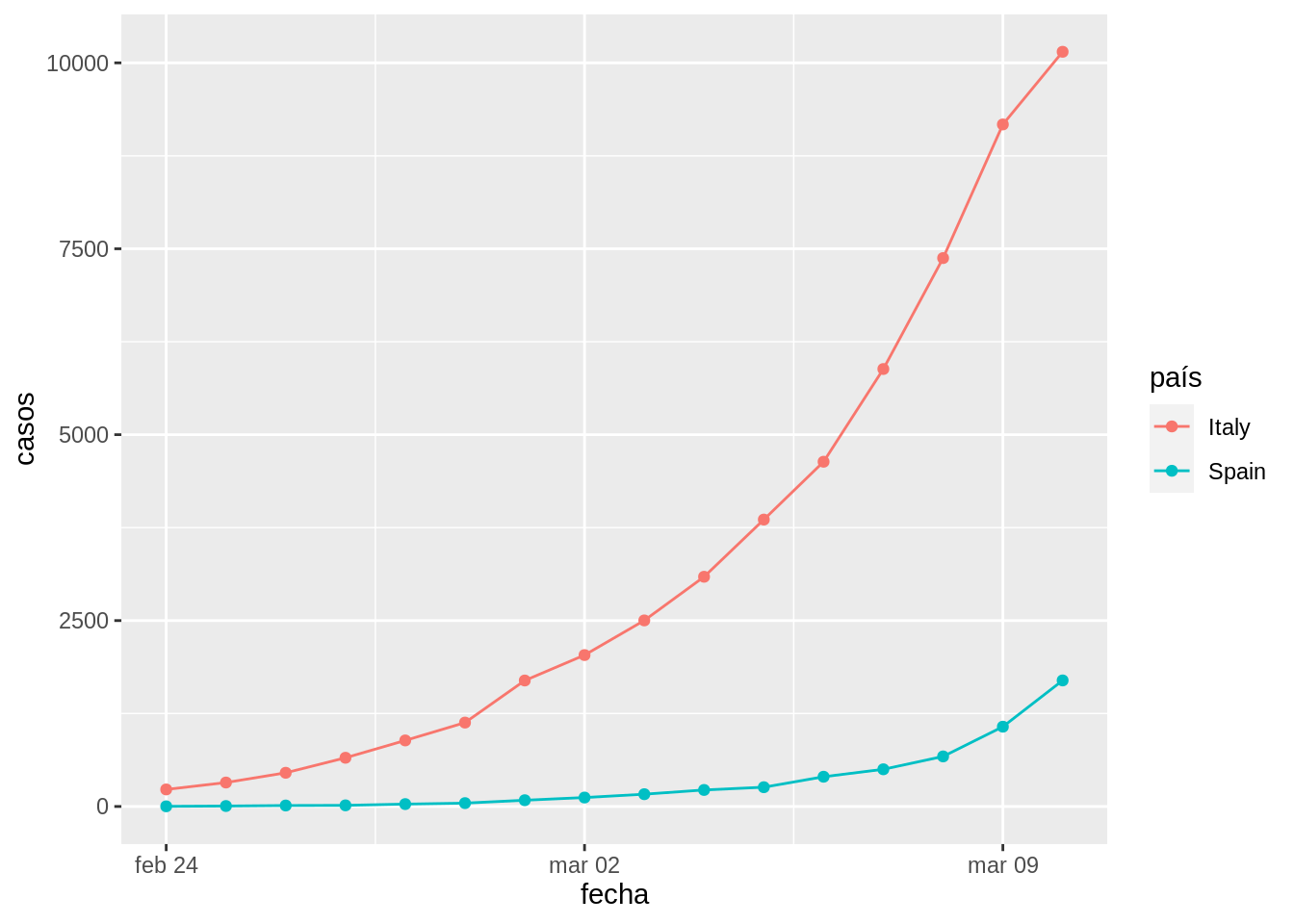
Si quitamos 9 días a la fecha de España vemos que estamso alineados a como estaba Itala en ese día.
tmp <- cvirus_it_sp
tmp$fecha[tmp$país == "Spain"] <- tmp$fecha[tmp$país == "Spain"] - 9
tmp %>%
filter(fecha > as.Date("2020-02-10")) %>%
ggplot(aes(x = fecha, y = casos, col = país)) +
geom_line(size = rel(1.2))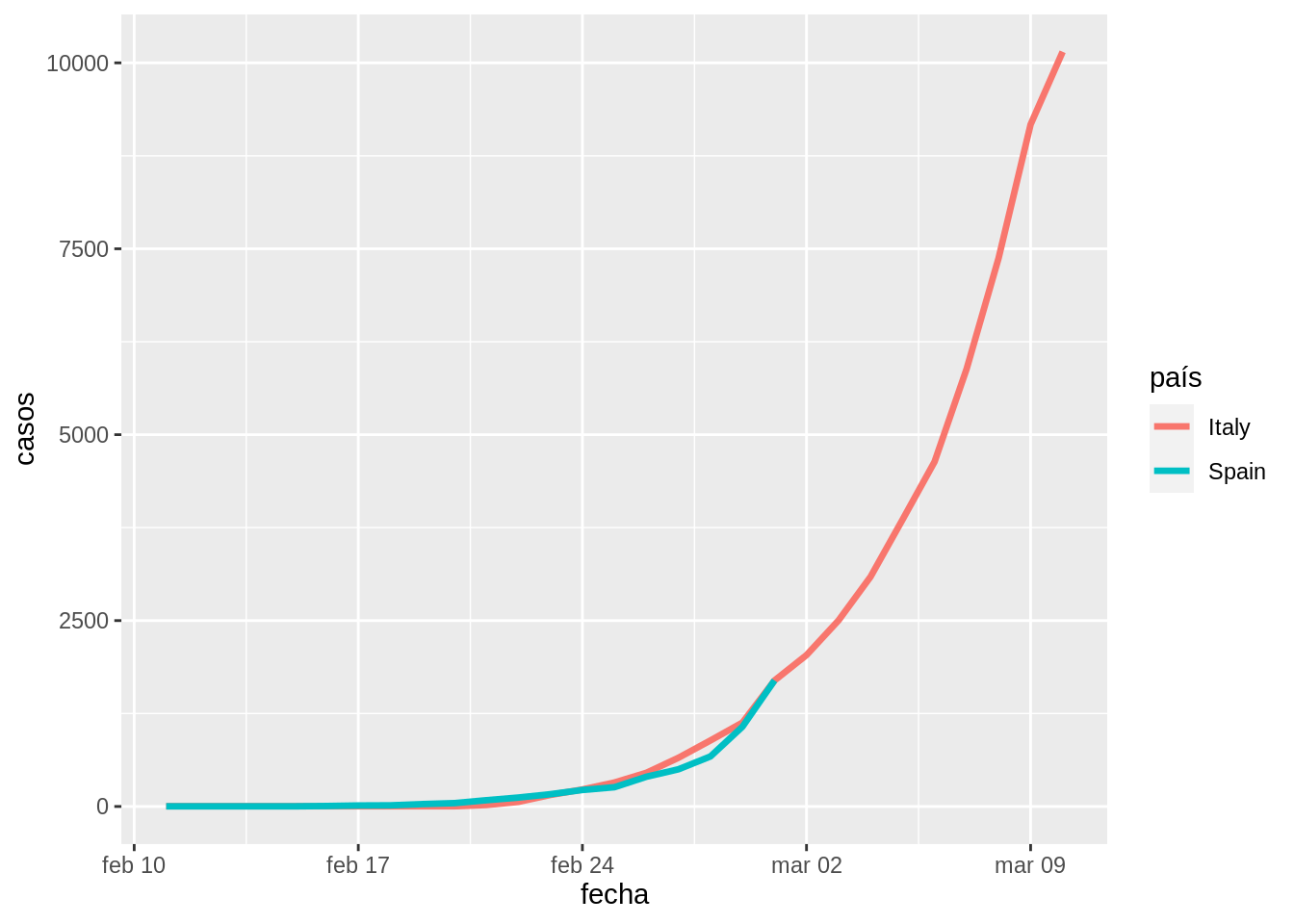
Y bueno, sigo plagiando a Carlos.
tmp <- tmp[tmp$fecha > as.Date("2020-02-10"), ]
ggplot(tmp, aes(x = fecha, y = log10(casos), col = país)) +
geom_line(size = rel(2))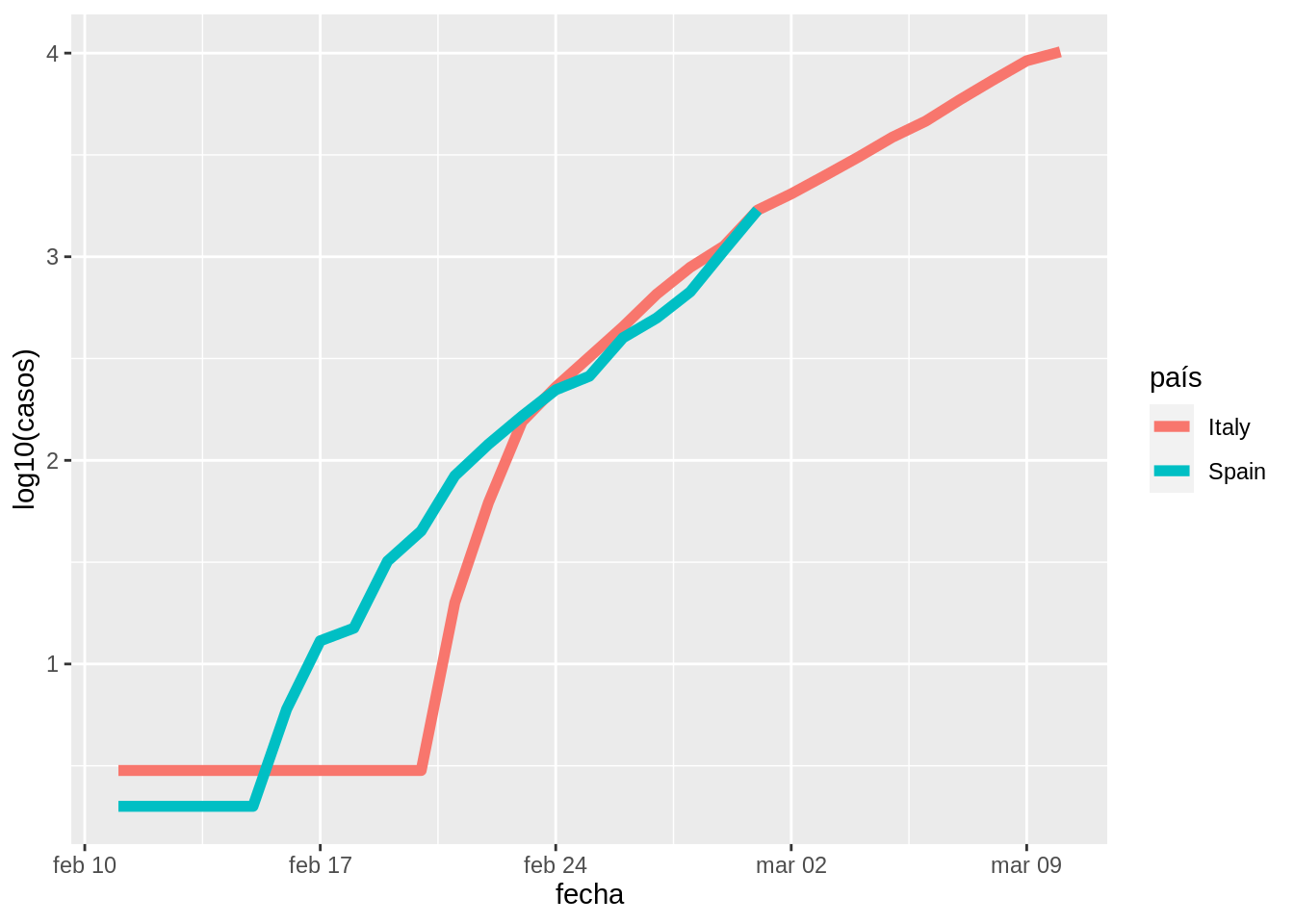
Mapa en leaflet
Y ahora un mapita con los datos del último día, del 9 de Marzo en el momento de escribir estas líneas
cvirus_map_data <- cvirus_longer %>%
filter(fecha == max(fecha))
pal <- colorNumeric(
palette = "Reds",
domain = c(-1, log(max(cvirus_map_data$casos + 1)))
)
leaflet(cvirus_map_data) %>%
# addProviderTiles('CartoDB.Positron') %>%
addProviderTiles("Stamen.Toner") %>%
addCircleMarkers(
lng = ~Long,
lat = ~Lat,
label = ~ paste0( país, ": ", casos ),
radius = ~ 3 * log( casos + 1 ) ,
color = ~ pal(log( casos + 1 ) )
) Y me he quedado con ganas de hacer un mapa con mapview del estilo de estos,
mapview, pero la verdad es que entre unas cosas y otras hoy ando bastante cansado. Mañana lo hago.