Qué es h2o
Aparte de lo que todos entendemos por h2o, h2o también es una empresa, y tiene algunos productos útiles para aquellos que nos dedicamos a lo que ahora se llama “ciencia de datos”. Más info aquí
h2o en R
El tema es que h2o tiene apis para usarlo con R, python, hadoop o maven. info
Veamos un pequeño ejemplo en R. En primer lugar instalamos h2o según las instrucciones. Lo que hace es bajarnos el paquete de R y también el artefacto que tiene h2o propiamente dicho. Se me olvidaba, h2o está hecho en Java.
# instalar
# The following two commands remove any previously installed H2O packages for R.
if ("package:h2o" %in% search()) { detach("package:h2o", unload=TRUE) }
if ("h2o" %in% rownames(installed.packages())) { remove.packages("h2o") }
# Next, we download packages that H2O depends on.
pkgs <- c("RCurl","jsonlite")
for (pkg in pkgs) {
if (! (pkg %in% rownames(installed.packages()))) { install.packages(pkg) }
}
# Now we download, install and initialize the H2O package for R.
install.packages("h2o", type="source", repos="http://h2o-release.s3.amazonaws.com/h2o/rel-xu/3/R")Levantamos el cluster de h2o, nótese que lo hago en local, pero también se puede instalar h2o y usarlo desde R en un cluster de máquinas.
library(h2o)
h2o.init(nthreads = 4, max_mem_size = "7G")Nos levanta también un UI muy bonito ( y que yo apenas uso) en el puerto 54321
Modelo de juguete
Vamos a hacer un modelo usando gradient boosting, que parece que no se usa otra cosa últimamente para modelar la altura en función del peso y del año de nacimiento de personajes de starwars.
library(tidyverse)## ── Attaching packages ─────────────────────────────────── tidyverse 1.2.1 ──## ✔ ggplot2 3.1.0 ✔ purrr 0.3.2
## ✔ tibble 2.1.1 ✔ dplyr 0.8.0.1
## ✔ tidyr 0.8.3 ✔ stringr 1.4.0
## ✔ readr 1.3.1 ✔ forcats 0.4.0## ── Conflicts ────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()df <- starwars %>% select(mass,height, hair_color,birth_year)Subimos los datos a h2o, a partir de esto el resto los cálculos los hace h2o y no R.
# Convertimos a datos de h2o
starwars_hframe <- as.h2o(df, destination_frame = "starwars_hframe")##
|
| | 0%
|
|=================================================================| 100%# train test 70 / 30
partition <- h2o.splitFrame(starwars_hframe, ratios = c(0.7), seed = 155)
# guardamos el conjunto de test en un csv para utlizarlo posteriormente.
write_csv(as.data.frame(partition[[2]]), path = "test.csv")Entrenamos el modelo y vemos algunas métricas
x = c("mass","birth_year")
y = "height"
mod_1 <- h2o.gbm(
model_id = "mi_gbm",
x = x,
y = y,
training_frame = partition[[1]])##
|
| | 0%
|
|=================================================================| 100%summary(mod_1)## Model Details:
## ==============
##
## H2ORegressionModel: gbm
## Model Key: mi_gbm
## Model Summary:
## number_of_trees number_of_internal_trees model_size_in_bytes min_depth
## 1 50 50 5459 2
## max_depth mean_depth min_leaves max_leaves mean_leaves
## 1 4 3.06000 4 5 4.14000
##
## H2ORegressionMetrics: gbm
## ** Reported on training data. **
##
## MSE: 467.436
## RMSE: 21.62027
## MAE: 14.87359
## RMSLE: 0.1437985
## Mean Residual Deviance : 467.436
##
##
##
##
##
## Scoring History:
## timestamp duration number_of_trees training_rmse
## 1 2019-03-17 19:44:47 0.041 sec 0 35.22147
## 2 2019-03-17 19:44:47 0.255 sec 1 33.70794
## 3 2019-03-17 19:44:47 0.276 sec 2 32.39930
## 4 2019-03-17 19:44:47 0.292 sec 3 31.32345
## 5 2019-03-17 19:44:47 0.307 sec 4 30.39768
## training_mae training_deviance
## 1 23.34364 1240.55202
## 2 22.17922 1136.22507
## 3 21.25695 1049.71432
## 4 20.58444 981.15835
## 5 20.12695 924.01906
##
## ---
## timestamp duration number_of_trees training_rmse
## 46 2019-03-17 19:44:48 0.662 sec 45 21.99193
## 47 2019-03-17 19:44:48 0.669 sec 46 21.91429
## 48 2019-03-17 19:44:48 0.675 sec 47 21.82580
## 49 2019-03-17 19:44:48 0.682 sec 48 21.75770
## 50 2019-03-17 19:44:48 0.689 sec 49 21.69123
## 51 2019-03-17 19:44:48 0.696 sec 50 21.62027
## training_mae training_deviance
## 46 15.16286 483.64494
## 47 15.11539 480.23606
## 48 15.03050 476.36561
## 49 14.98415 473.39766
## 50 14.91394 470.50961
## 51 14.87359 467.43601
##
## Variable Importances: (Extract with `h2o.varimp`)
## =================================================
##
## Variable Importances:
## variable relative_importance scaled_importance percentage
## 1 mass 218099.125000 1.000000 0.947798
## 2 birth_year 12012.344727 0.055077 0.052202mod_1 %>% h2o.performance(newdata = partition[[2]])## H2ORegressionMetrics: gbm
##
## MSE: 772.518
## RMSE: 27.79421
## MAE: 21.01117
## RMSLE: 0.1946992
## Mean Residual Deviance : 772.518En la UI localhost:54321 se muestra todo esto de manera más “bonita”.
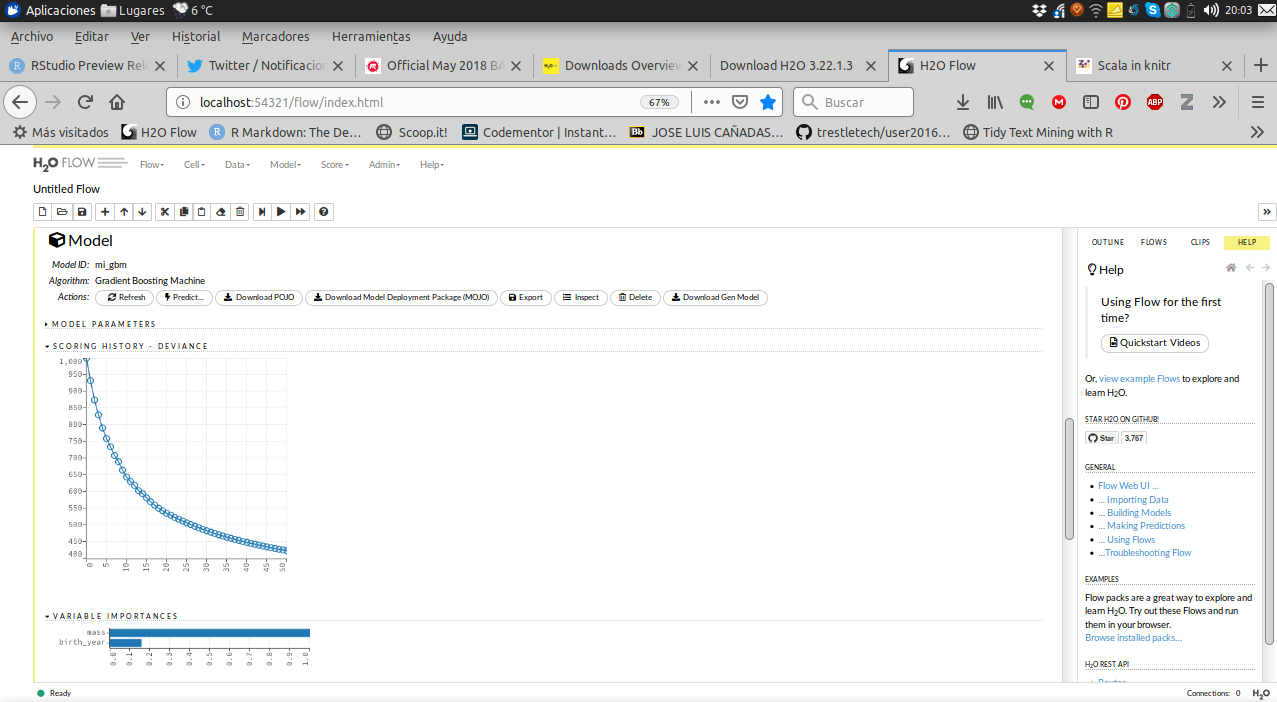
Predicción del modelo
Pues muy fácilmente
mod_1 %>% h2o.predict(partition[[2]])##
|
| | 0%
|
|=================================================================| 100%## predict
## 1 201.8132
## 2 117.8270
## 3 177.1644
## 4 175.6902
## 5 176.2006
## 6 201.8132
##
## [26 rows x 1 column]Todo esto está muy bien, pero diréis, ¿cómo pongo esto en un entorno productivo?
Productivizar modelos h2o
H2O permite exportar los modelos como una cosa que ellos llaman MOJO. Básicamente lo que se generan son dos archivos, un artefacto .jar que tiene las librerías, clases y métodos de java necesarios para predecir y un archivo .zip dónde está las especificaciones del modelo, en este caso los árboles.
Con estos dos ficheros ya se puede utilizar ese modelo para predecir en cualquier sitio dónde haya JAVA e incluso crear UDFS en HIVE y , por supuesto también se pueden utilizar con spark.
Exportar modelo
Descargar el archivo zip y el jar es bastante sencillo.
h2o.download_mojo(mod_1, get_genmodel_jar = TRUE,
genmodel_name = "mojo_jar.jar")## [1] "mi_gbm.zip"Paramos h2o.
h2o.shutdown(prompt = FALSE)## [1] TRUEPredecir en spark
Ahora podemos usar el jar y el zip para productivizar nuestro modelo en un cluster de spark sin necesidad de tener h2o instalado.
Lanzamos un spark-shell añadiendo el .jar para que lo distribuya.
# lo lanzo en mi spark instalado en local pero también sirve en un cluster con yarn
~/spark/spark-2.4.0-bin-hadoop2.7/bin/spark-shell --conf spark.driver.memory="2g" --conf spark.executor.memory="2g" --conf spark.executor.instances=2 --conf spark.executor.cores=2 --jars mojo_jar.jar
Pues ya podemos predecir nuestro modelo en spark de la siguiente forma
import _root_.hex.genmodel.GenModel
import _root_.hex.genmodel.easy.{EasyPredictModelWrapper, RowData}
import _root_.hex.genmodel.easy.prediction
import _root_.hex.genmodel.MojoModel
import _root_.hex.genmodel.easy.RowData
import org.apache.spark.sql.Column
// cargar mi modelo
val modelPath = "mi_gbm.zip"
// Cargar datos de test para predecir
val dataPath = "test.csv"
// Import data
val dfStarWars = spark.read.option("header", "true").
csv(dataPath)
// Import MOJO model
val mojo = MojoModel.load(modelPath)Instanciamos la clase EasyPredictModelWrapper
val easyModel = new EasyPredictModelWrapper(
new EasyPredictModelWrapper.Config().
setModel(mojo).
setConvertUnknownCategoricalLevelsToNa(true).
setConvertInvalidNumbersToNa(true))
El modelo en MOJO y las clases que ha generado h2o necesitan que pasemos de SparkDataframe a rowData. Una forma fácil de hacerlo es hacer un map sobre todas las columnas. Una vez convertida al formato adecuado, dentro del map se utiliza el método predictRegression para obtener la predicción del modelo y con .toDF lo pasamos a sparkdataframe. Ojo, EasyPredictModelWrapper usa las variables como String, pero internamente ya sabe si son numéricas o no.
// -------------
// Convertir todas las columnas a rowdata
// -------------
val header = dfStarWars.columns
val dfScore = dfStarWars.map {
x =>
val r = new RowData
header.indices.foreach(idx => r.put(header(idx), x.getAs[String](idx) ))
val score = easyModel.predictRegression(r).value
(score)
}.toDF("predict")
Y lo que obtenemos es un sparkdataframe que podremos guardar en una tabla de hive o seguir utilizando dentro de la sesión de spark
Comprobamos que la predicción es la misma de antes.
dfScore.show()
+------------------+
| predict|
+------------------+
|201.81315093098914|
|117.82702476608341|
| 177.1644296299923|
|175.69017681556528|
| 176.200564921673|
|201.81315093098914|
|176.56103480311816|
|179.59632814752644|
|118.87230888831203|
| 182.1062176607806|
|172.80586874375825|
|179.59632814752644|
|179.59632814752644|
|202.99927356171196|
|149.69601927029674|
|179.59632814752644|
|150.02756194102352|
| 183.5801466245461|
|181.64131501207834|
|179.59632814752644|
+------------------+
only showing top 20 rows
El siguiente paso sería en vez de ejecutar un spark-shell convertir todo en una aplicación de Spark, pero para eso necesitaré a mis queridos “ingenazis”.